Speicherung und Prozessierung von Daten
Allgemeine Empfehlungen für die Speicherung von Forschungsdaten sind:
- Namenskonvention: Verwenden Sie kurze, aussagekräftige Datei- und Ordnernamen zur einfachen Identifizierung.
- Ordnerstruktur: Organisieren Sie die Daten logisch in einer klaren, einheitlichen Ordnerstruktur.
- Datenformate: Wählen Sie offene, dokumentierte, von der Fachcommunity empfohlene Standardformate, um langfristige Lesbarkeit zu gewährleisten.
- Metadaten: Erfassen Sie ausreichend standardisierte Metadaten, um Interpretierbarkeit und Reproduzierbarkeit zu ermöglichen.
- Checksummen: Berechnen Sie Checksummen (z.B. MD5 oder SHA256) für die Dateien zur Überprüfung der Datenintegrität.
- Versionierung: Nutzen Sie ein Versionierungssystem (z.B. Git) zur Nachverfolgung von Änderungen an Daten und Metadaten.
- Speicherort: Wählen Sie ein Langzeitarchivierungssystem, das die Datenintegrität und Zugänglichkeit dauerhaft gewährleistet.
Checksumme erstellen
1. Drücken Sie Windows+R, um das Fenster Ausführen zu öffnen.
2. Geben Sie cmd ein und klicken Sie auf OK.
3. Das Command Prompt Fenster öffnet sich.
4. Führen Sie folgenden Befehl aus:
certutil -hashfile C:\pfad\meine_datei.tif SHA256
Info: Ein Pfad mit Leerzeichen muss in Anführungszeichen gesetzt werden.
Format für Speicherung und Langzeitarchivierung
In der biologischen Forschung fallen vielfältige Daten aus Geräten wie Mikroskopen, Sequenzierern oder Massenspektrometern an. Jedes dieser Instrumente verwendet häufig eigene (proprietäre) Datenformate, die oft nur mit spezieller Software lesbar sind. Dies erschwert jedoch enorm den Datenaustausch, die Weiterverarbeitung der Daten sowie die langfrisitge Nutzung bzw. Langzeitarchivierung der Daten.
In solchen Fällen empfehlen wir eine zusätzliche Konvertierung der Daten in ein gängiges Datenformat (Standardformat). Standardformate zeichnen sich durch öffentlich zugängliche Spezifikationen, Herstellerunabhängigkeit und breite Softwarekompatibilität aus.
Standardformate für Mikroskopiedaten
Für die Speicherung und Archivierung von Mikroskopiedaten nutzen Sie bitte die gängigsten Datenformate. Vermeiden Sie die proprietären Formate der Hersteller, sowie JPEG oder PNG. Nutzen Sie stattdessen:
OME-TIFF: Empfohlen, wenn OME-XML-Metadaten direkt in der Bilddatei gespeichert werden sollen.
- Vorteile: Standardisierte Metadatenstruktur (OME-XML), optimiert für große Bilddatensätze, unterstützt mehrdimensionale Daten, weit verbreitet.
- Nachteil: Nicht optimaler S3-Zugriff.
OME-Zarr: Objektbasiertes Format für große Datensätze und Cloud-basierte Workflows.
- Vorteile: Kombination aus OME-XML-Metadaten und effizienter Speicherung für schnellen Zugriff - egal ob lokal oder online ("in der cloud"), Skalierbarkeit, S3-optimiert (z.B. Amazon Simple Storage Service bzw. vergleichbare Objektspeicher), paralleler Zugriff, Unterstützung für große Datensätze und Metadaten.
- Nachteil: Nicht optimal für sehr kleine Datensätze. Zerlegung in viele kleine Dateien kann zu Problemen auf klassischen Festplatten und Dateisystemen führen.
HDF5 (Hierarchical Data Format version 5): Geeignet für sehr große Datensätze und mehrdimensionale Daten.
- Vorteile: Effiziente Speicherung großer Datenmengen, unterstützt Metadaten, flexibel.
- Nachteil: Nicht optimaler S3-Zugriff.
Möglichkeiten der Speicherung von Metadaten
Metadaten geben Auskunft über Entstehung, Inhalt und die Nachnutzung von Forschungsdaten. Sie werden oft als „Daten über Daten“ bezeichnet und sind entscheidend für die Qualität und Integrität der Forschungsdaten. Obwohl sie eigenständige Daten darstellen, werden sie häufig gemeinsam mit den eigentlichen Forschungsdaten gespeichert.
Für die Metadaten-Speicherung gibt es unterschiedliche Möglichkeiten:
- Eingebettete Metadaten: Speicherung von Metadaten direkt in der Datendatei (z.B. Bild).
- Sidecar Metadaten: Speicherung von Metadaten in seperaten Dateien.
- Datenbanken: Speicherung von Metadaten in einer entsprechenden Datenbank (z.B. OMERO, openBIS), die mit den Daten verknüpft ist.
Nutzung fachspezifischer Metadatenstandards
Metadaten umfassen technische und fachliche Daten. Technische Metadaten (z.B. Datenvolumen, Datenformat) spielen bei der langfristigen Datenspeicherung eine Rolle. Fachliche Metadaten (z.B. Fachbegriffe, domänenspezifisches Wissen oder Kontextinformationen) gewährleisten die Verständlichkeit und Nachnutzung der Forschungsdaten.
Die spezifischen Anforderungen an Metadaten variieren je nach Fachdisziplin und werden in sogenannten fachspezifischen Metadatenstandards festgelegt, die sich im Umfang, Vokabular, Struktur und/oder Format unterscheiden können.
Tipps
- Wenn möglich, sollten Normdaten und kontrollierte Vokabulare oder Ontologien zur Beschreibung der Daten verwendet werden.
- Eine frühzeitige Erfassung von Metadaten verringert den Dokumentationsaufwand zum Ende des Projektes.
- Repositorien und Journals können spezifische Anforderungen an die zu archivierenden Datensätze und ihre Metadaten haben.
Wichtige Metadatenstandards in der biologischen Forschung
Bilddaten:
- OME-XML (Open Microscopy Environment XML): Beschreibung der technischen Metadaten eines bildgebenden Experiments
- REMBI (Recommended Metadata for Biological Images)
- MITI (Minimum Information for Highly Multiplexed Tissue Imaging)
- MIHCSME (Minimum Information for High Content Screening Microscopy Experiments)
Sequenzdaten:
- FASTA/ FASTQ: Standardformate für DNA-/RNA-Sequenzen und zugehörige Qualitätsdaten.
- SAM/ BAM/ CRAM: Für alignierte Sequenzdaten.
- MIxS/MIMARKS: Mindestmetadaten-Standards für verschiedene Arten von Sequenz- und Metadaten.
Bioinformatik- und Omics:
- MIAME (Minimum Information About Microarray Experiments)
- MINSEQE (Minimum Information about high throughput Sequencing Experiments)
- ISA-Tab / ISA-JSON: Framework für komplexe experimentelle Studien (z.B. Omics-basierte Studien).
Bitte nutzen Sie für die Speicherung und Archivierung von digitalen Forschungsdaten die von der IT des Fachbereichs Biologie/Chemie bereitgestellte Infrastruktur:
Alternativ bzw. zusätzlich nutzen Sie bitte immer nur anerkannte nationale oder internationale (fachspezifische) Repositorien oder Archive.
Die Aufbewahrungsfrist für Forschungsdaten und Aufzeichnungen beträgt mindestens zehn Jahre entweder nach Veröffentlichung der Forschungsdaten oder ab Veröffentlichung der Forschungsergebnisse oder nach Abschluss der jeweiligen Forschungstätigkeit. Abweichungen können sich aus gesetzlichen oder vertraglichen Vorschriften, aus Vorgaben von Drittmittelgebern oder internen Richtlinien ergeben.
Weitere offene Angebote
GitLab - Repositorium für Softwarecode (Universität Osnabrück)
Chemotion - Elektronisches Laborbuch für Chemiker (Universität Braunschweig)
Eine Software-gestützte Prozessierung und Analyse von Forschungsdaten ermöglicht zwar die Auswertung dieser Daten, aber auch dieser Prozess erfordert ein umfassendes Datenmanagement, einschließlich Metadaten, Versionierung und sicherer Archivierung, um die Reproduzierbarkeit und Nutzbarkeit der Forschungsdaten zu gewährleisten.
Tipps:
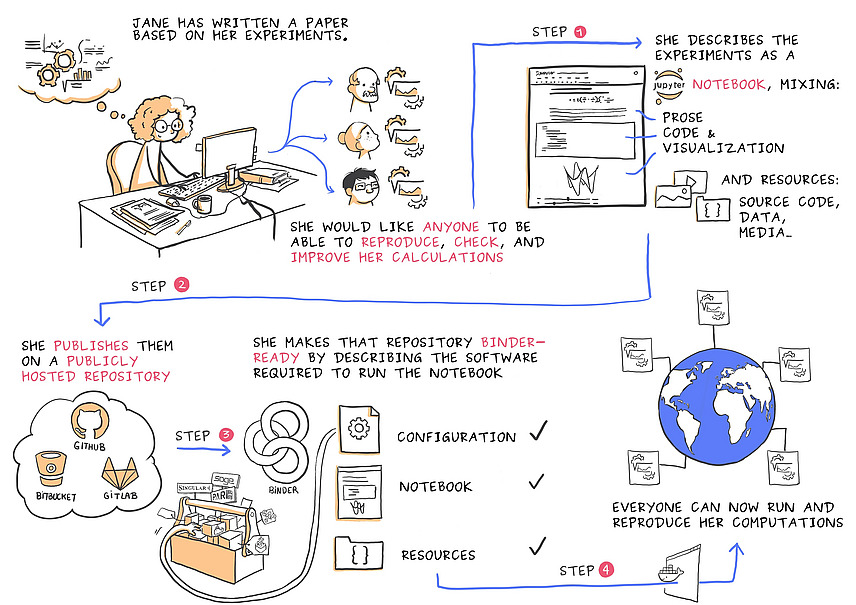
- Nutzen Sie für Bilddaten eine direkte Anbindung von Fiji/ImageJ und OMERO, um Ihre Ergebnisse automatisch zu verlinken.
- Nutzen Sie Tools wie Jupyter Notebook oder den Macrorecorder von Fiji, die neben einer Automatisierung auch eine reproduzierbare Dokumentation Ihres Analyseworkflows gewährleisten, und verlinken Sie diese mit Ihren Daten.